Genética Molecular Planta-bacteria
GENÉTICA MOLECULAR DE INTERACCIONES PLANTA-BACTERIA
TITULO: Interacción de bacterias fitopatógenas con plantas leñosas
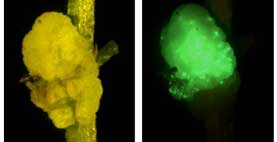
Se utiliza como modelo la interacción de la bacteria patógena Pseudomonas savastanoi pv. savastanoi (Psv) con su hospedador natural, el olivo. La secuenciación en 2010 del borrador del genoma de la cepa Psv NCPPB 3335, así como la identificación de nuevos genes de virulencia de este patógeno mediante la utilización de la estrategia de análisis genómico funcional “Signature tagged mutagenesis” (STM) nos ha permitido establecer a esta bacteria patógena perteneciente al complejo Pseudomonas syringae en un modelo para el análisis molecular de interacciones entre bacterias patógenas y plantas leñosas.
RESPONSABLE/S: Cayo Ramos Rodríguez y Luis Rodriguez Moreno
Miembros:
Antonio Arroyo Mateo, Hilario Domínguez Cervan, Isabel Imbroda Solano, Carla A. Lavado Benito, Alba Moreno Pérez, Adrian Pintado Calvillo
Proyectos Activos:
- Título: Análisis posgenómico del repertorio de virulencia de Pseudomonas savastanoi: huéspedes leñosos vs. herbáceos (SAVAS.v5)
- Duración: desde el 01/09/2021 hasta el 31/08/2024
- Entidad financiadora: Plan Nacional I+D+i Ministerio de Ciencia e Innovación, Cofinanciado por el Fondo Europeo de Desarrollo Regional (FEDER)
- Investigadores principales: Cayo Ramos Rodríguez y Luis Rodríguez Moreno
- Participantes (UMA): Antonio Arroyo Mateo, Hilario Domínguez Cerván, Isabel Imbroda Solano, Carla Lavado Benito, Alba Moreno Pérez, Adrián Pintado Calvillo.
- Participantes externos: Leo Eberl (University of Zurich, Suiza), Vittorio Venturi (International Centre for Genetic Engineering and Biotechnology-ICGEB, Trieste, Italia).
-Resumen: Los patógenos de plantas reducen el rendimiento y la calidad de la producción agrícola, así como la seguridad alimentaria. El control de los patógenos generalmente requiere la utilización de plaguicidas químicos dañinos para las personas, animales y el medio ambiente, y no se prevé disponer de alternativas de control seguras y efectivas en un futuro próximo. La investigación más actual se dirige al entendimiento de las bases moleculares de la patogenicidad y la definición del espectro de huésped. Este conocimiento es esencial para el desarrollo de nuevas estrategias de manejo de enfermedades que minimicen la utilización de pesticidas químicos, y exploten la resistencia vegetal como un método de control económico, eficaz y respetuoso con el medio ambiente. Los patovares de Pseudomonas savastanoi de huéspedes leñosos (olivo, adelfa, fresno, retama y dipladenia) y el patógeno de judía P. savastanoi pv. phaseolicola (Pph) son modelos bien establecidos para el estudio de los determinantes moleculares de la patogenicidad de plantas leñosas y herbáceas. Además, su proximidad filogenética y su rango de huésped divergente los convierten en candidatos ideales para investigar las bases moleculares que definen el rango de huésped, tanto a nivel de patovares como de razas. El análisis de genómica comparativa y funcional de este grupo de bacterias, está facilitando la identificación de los determinantes de patogenicidad que podrían participar en la definición del rango de huésped y en la evolución de la virulencia en este complejo bacteriano. Esta definición del espectro de huésped y la virulencia, sin embargo, dependen de un variado conjunto de determinantes genéticos que es necesario estudiar desde distintas perspectivas, que incluyen el estudio de i) el papel de los efectores de tipo III y sus variantes alélicas en la definición del espectro de huésped, ii) el grado de expresión de genes de virulencia y su regulación diferencial mediante diversos sistemas de regulación global y, iii) la identificación y caracterización del secretoma extracelular mediante vesículas de membrana (VMs), y de su papel en la virulencia. En consecuencia, nuestra hipótesis de trabajo es que “la especificidad de huésped y la virulencia de los patovares y razas de P. savastanoi implica la acción coordinada de diversos determinantes genéticos, y de sus alelos, cuya expresión está modulada por una compleja red de redes de regulación interconectadas”. Como continuación de nuestros proyectos anteriores en esta línea, se proponen los siguientes objetivos: 1) Evaluar el papel del regulón HrpL en la patogenicidad y definición de razas, y 2) Caracterizar la termorregulación y las redes de regulación y su impacto en la virulencia. Como una nueva aproximación para evaluar la hipótesis de trabajo, se propone el objetivo 3) Identificar y caracterizar el catálogo de moléculas liberadas a través de sistemas de secreción y vesículas de membrana (VMs), así como determinar su papel en el desarrollo de enfermedad. Los resultados de este proyecto se dirigen a ampliar el conocimiento de las interacciones entre patógenos bacterianos y sus huéspedes leñosos y herbáceos, así como a contribuir al desarrollo de métodos de control de enfermedades bacterianas de plantas mediante una mejor identificación y manejo de los patovares, razas y patógenos emergentes.
Título: El secretoma de la bacteria fitopatógena Pseudomonas savastanoi: Identificación de nuevas proteínas extracelulares y papel en virulencia durante su interacción con el olivo. (SAVASTONI-SECRET)
- Duración: desde el 01/09/2021 hasta el 31/12/2022
- Entidad financiadora: Consejería de Transformación Económica, Industria, Conocimiento y Universidades de la Junta de Andalucía, Subvenciones PAIDI-2020, Cofinanciado por el Fondo Europeo de Desarrollo Regional (FEDER)
- Investigadores principales: Luis Rodríguez Moreno
- Participantes (UMA): Cayo Ramos Rodríguez, Antonio Arroyo Mateo, Hilario Domínguez Cerván, Isabel Imbroda Solano, Carla Lavado Benito, Alba Moreno Pérez, Adrián Pintado Calvillo.
- Participantes externos: Vittorio Venturi (International Centre for Genetic Engineering and Biotechnology-ICGEB, Trieste, Italia); Emilia López Solanilla (Centro de Biotecnología y Genómica de Plantas-CBGP, Universidad Politécnica de Madrid, Madrid, España); Jesús Murillo Martínez (Institute for Multidisciplinary Research in Applied Biology, Universidad Pública de Navarra, Pamplona, España).
-Resumen: La comunidad andaluza es la región española con una mayor superficie dedicada al cultivo del olivo con más de un millón y medio de hectáreas, y con una actividad industrial que cohesiona a más de 250.000 familias. Sin embargo, este cultivo resulta afectado por un gran número de plagas y patógenos de origen fúngico y bacteriano que pueden afectar la producción y calidad del fruto. Uno de los gastos más cuantiosos generados en las explotaciones olivareras resulta del tratamiento fitosanitario para prevenir las enfermedades de origen fúngico, ya que no existen tratamientos preventivos eficaces para evitar las enfermedades bacterianas, y los tratamientos químicos disponibles se reducen en la práctica a los compuestos cúpricos, cuya eficacia es generalmente moderada. Por tanto, el desarrollo de estrategias novedosas de control de enfermedades del olivar requiere necesariamente de la identificación de los factores necesarios para la supervivencia y virulencia de los patógenos. Aunque en la actualidad existe cierto nivel de conocimiento sobre los factores que determinan las enfermedades bacterianas en plantas herbáceas, dichos factores permanecen prácticamente desconocidos para las enfermedades de plantas leñosas. El objetivo principal de este proyecto es la identificación de factores de virulencia, no dependientes del sistema de secreción tipo III, durante la interacción P. savastanoi olivo. Para ello se utilizará un abordaje experimental basado en una aproximación proteómica utilizando una cepa silvestre de P. savastanoi (NCPPB 3335) y un mutante afectado en el sistema de secreción tipo III (NCPPB 3335-DhrpA) en dos condiciones experimentales distintas: i) plantas de olivo in vitro; ii) medio de inducción hrp. Las proteínas identificadas se utilizarán para identificar factores de virulencia homólogos en otras bacterias fitopatógenas, que pudieran servir para la creación de estrategias de control efectivas que prevengan las infecciones bacterianas en huéspedes leñosos.