Profundizan en el ‘mapa oculto’ de las proteínas y abren la puerta a nuevas terapias para enfermedades raras
 1
/
1
1
/
1
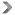
La herramienta, desarrollada por el grupo SIBIUMA, avanza en la comprensión y el tratamiento de estas patologías
Categoría: Investigación, portada
Un equipo científico formado por investigadores del grupo Bases Moleculares de los Sistemas Biológicos (SIBIUMA) de la Universidad de Málaga, también pertenecientes a IBIMA Plataforma BIONAND, del Centro de Investigación Biomédica en Red de Enfermedades Raras (CIBERER) y del Instituto Nacional de Bioinformática (INB/ELIXIR-ES) de España, ha dado un gran paso para ayudar en la comprensión de las enfermedades raras, abriendo la puerta a nuevas estrategias diagnósticas y terapéuticas.
El estudio, que ha sido publicado en la revista científica ‘Briefings in Bioinformatics’, introduce una potente metodología para analizar las complejas interacciones entre proteínas en el cuerpo humano.
“Imagina que las proteínas de nuestro cuerpo son como personas en una inmensa red social, y sus interacciones son las ‘amistades’. Para entender qué grupos de proteínas trabajan juntas en funciones biológicas o enfermedades, es crucial identificar sus ‘comunidades’. El desafío es que, al igual que una persona puede pertenecer a varios grupos de amigos, una proteína puede estar involucrada en múltiples procesos biológicos simultáneamente”, explica el investigador del Departamento de Biología Molecular y Bioquímica James Richard Perkins, uno de los autores de este trabajo.
Los métodos tradicionales solían asignar cada proteína a una única comunidad, simplificando demasiado la realidad y perdiendo información valiosa, según este investigador. Para superar esto, los investigadores han desarrollado una "interesante estrategia que imita cómo una persona puede formar parte de varios círculos sociales a la vez".
Aspectos claves del avance
La clave del avance radica en la creación de un nuevo ‘mapa’ biológico teniendo en cuenta los múltiples ‘círculos sociales’ de cada proteína, una herramienta innovadora que permite representar las redes de interacción entre proteínas en un espacio matemático mucho más simple e intuitivo. Esta tecnología, tal y como señala Perkins, actúa como si transformáramos un complejo mapa de carreteras en otro donde las ciudades —en este caso, las proteínas— con funciones similares se agrupan más cerca entre sí. Este enfoque facilita la detección de relaciones ocultas y abre nuevas posibilidades para comprender cómo se organizan y colaboran estas moléculas en los procesos celulares.
Para poder realizar esto, los científicos han mejorado significativamente un algoritmo conocido como Hierarchical Link Clustering (HLC), capaz de identificar comunidades de proteínas que se solapan. Esta capacidad resulta esencial, ya que, “en la biología real, al igual que en la vida social, un mismo elemento puede formar parte de distintos grupos”. “Así como una persona puede pertenecer a varios círculos de amigos, una proteína puede estar implicada en distintas funciones biológicas, y esta metodología permite capturar precisamente esa complejidad”, afirman.
Por último, han logrado optimizar la representación de las vías biológicas conocidas al restringir los ‘paseos aleatorios’ del algoritmo dentro de estas comunidades definidas por HLC. “Esto ha mejorado de forma drástica la precisión del mapa digital: ahora, las proteínas que participan en la misma ruta biológica tienden a aparecer mucho más próximas entre sí. Esta mejora en la navegación del mapa permite estudiar de manera más clara y eficiente las interacciones y funciones de los sistemas biológicos”, concluye el científico de la UMA.

Parte del equipo científico de SIBIUMA en el jardín Botánico de la Universidad, frente a la Facultad de Ciencias
Impacto directo en las enfermedades raras: El caso de las RASopatías
La metodología ha sido aplicada a un grupo de enfermedades raras interrelacionadas conocidas como RASopatías. Estas condiciones, causadas por mutaciones en genes de una vía clave (RAS/MAPK), presentan una amplia gama de síntomas. A pesar de los avances en genética, los mecanismos completos que las impulsan y las conexiones entre ellas no están del todo claros.
Gracias a este nuevo enfoque, los investigadores no solo han logrado representar mejor las vías conocidas de las RASopatías, sino que también han podido identificar nuevos genes candidatos que podrían estar asociados con estas enfermedades, incluyendo síndromes como el de Noonan y Costello. La elección de este grupo de enfermedades se debe a la participación del grupo de investigación de la UMA en un proyecto europeo sobre este conjunto de enfermedades, el proyecto EURAS, en el que se pretende estudiarlas y desarrollar terapias específicas.
¿Qué significa esto para el futuro?
Este avance abre la puerta a "importantes aplicaciones biomédicas". Por un lado, al comprender mejor cómo interactúan los genes y cómo se organizan las rutas biológicas, se identifican posibles nuevas dianas terapéuticas, fundamentales para el desarrollo de fármacos más precisos y eficaces.
Por otro, el estudio revela una interesante conexión molecular entre las RASopatías y ciertos tipos de cáncer. Esta coincidencia sugiere que tratamientos oncológicos ya disponibles podrían reutilizarse para abordar estas patologías minoritarias, lo que representa una esperanza concreta para muchos pacientes.
Además, los autores han puesto a disposición de la comunidad científica las herramientas optimizadas utilizadas en este trabajo, fomentando así la colaboración y la aplicación de estos métodos en otros contextos biomédicos.
En conjunto, este “logro” —resultado de la cooperación entre destacadas instituciones de investigación— representa un paso decisivo hacia la comprensión profunda de las redes biológicas. Y, lo que es más importante, “ofrece un futuro más prometedor para el diagnóstico y tratamiento de enfermedades que hasta ahora planteaban enormes desafíos médicos y científicos”.
Referencia del estudio:
Federico García-Criado, Pedro Seoane, Elena Rojano, Juan A G Ranea, James R Perkins, Advancing edge-based clustering and graph embedding for biological network analysis: a case study in RASopathies, Briefings in Bioinformatics, Volume 26, Issue 4, July 2025, bbaf320, https://doi.org/10.1093/bib/bbaf320





